アプリケーションノート
Vol.4 | DNAチップと定量PCRの検出相関性
DNAチップは数百から数十万に上る網羅的な遺伝子を、少量の検体と比較的短時間で検出することができ、多くの生物学的研究に利用されている。米国FDAはDNAチップを用いた研究の質をさらに向上させるために、2006年からMAQC(MicroArray Quality Control)プロジェクト1)を主導し、7社のプラットフォームを用いて、DNAチップによる発現遺伝子検出の妥当性を検討した。その結果、プラットフォームの違いによらず、DNAチップによる検出結果の標準化はおおむね可能であると結論づけられている。
一方、現在もなお、DNAチップデータとqRT-PCRとのデータ相関性の不足が課題として挙げられることが多い。実はMAQCにおいてもTaqMan法との相関性が評価項目に挙げられているが、いずれのプラットフォームにおいても相関が低い遺伝子が散見される。
この現象の原因としてプローブ設計、ハイブリ条件の特異性等が考えられるが、我々は検出されるべきmRNAの前処理工程に注目した。すなわち、現在ほとんどのDNAチップ検出プロトコールでは、T7プライマーを利用した非特異的なRNA転写によって増幅したRNAを検出しているが、すべての遺伝子が均等に増幅される保証はなく、このバイアスがqRT-PCRとの違いをもたらしている可能性がある。しかし、mRNAは一般にtotal RNAの5%に過ぎず、限られた検体量中のmRNAを増幅することなく検出することは難しい。
そこで我々は、mRNAを増幅することなく検出することができる「非増幅法」を3D-Gene®と組合せて開発し、その検出結果をqRT-PCR法および増幅法と比較した。非増幅法では、mRNAをcDNAに逆転写したものを直接DNAチップ上で検出するため増幅バイアスを避けることができるが、シグナルが微小なため高いプラットフォームの感度が要求される。3D-Gene®を用いた検出の結果、非増幅法のqRT-PCRとのデータ相関性は予想通り、増幅法に比べて向上したことが示された(図1,2)。3D-Gene®と今回開発した高感度な非増幅法の組み合わせは、さらに定量性の高い実験への適応のみならず、バリデーションが重要であり、検出コスト削減が要求される検査・診断用途DNAチップの開発にも貢献することが可能である。
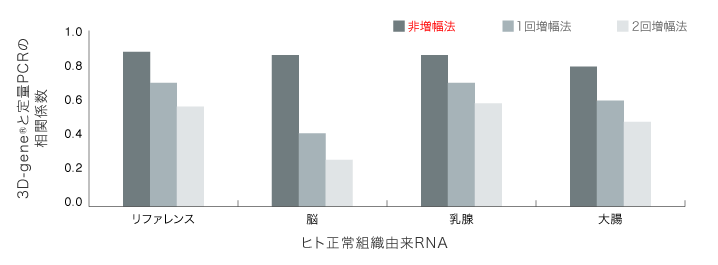
図1 定量RT-PCRとDNAチップの相関性
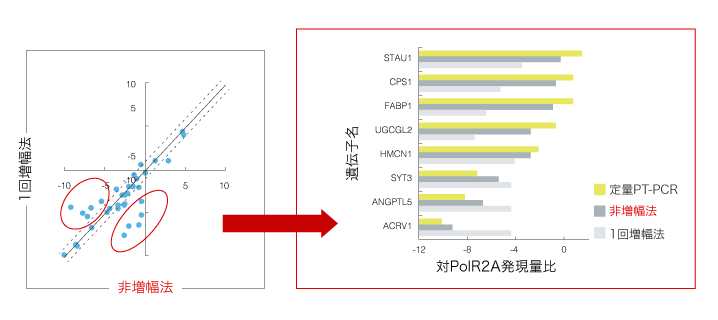
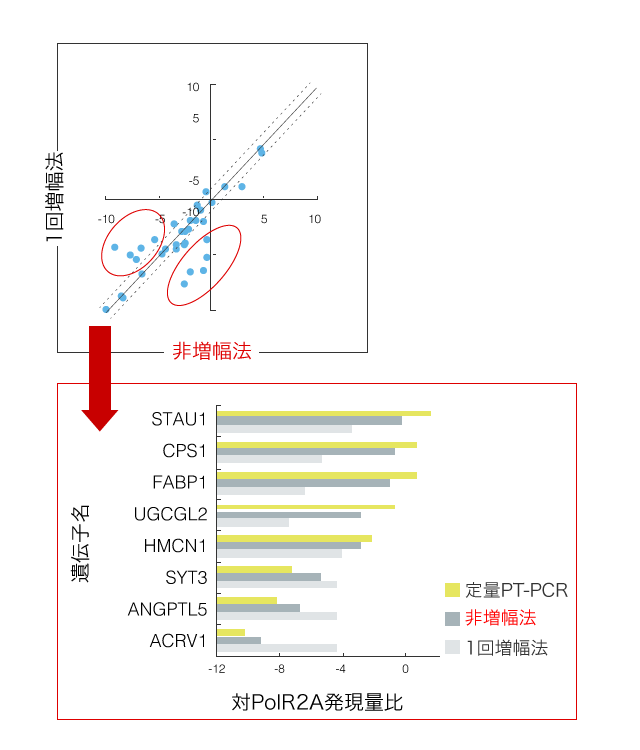
図2 非増幅法と増幅法の検出結果比較
参考論文:
- 1) Nature Biotechnology 24(9),2006.




